主要使用陳凱儀 老師lab的protocol
1.2 限制酶切割
使用Cut Smart Buffer,37°C培養,使用PCR機。
1.3 P1 Adapter連結酶反應
1.4 樣本合併
一次震碎體積為100uL,一次可震12個合併樣本。40個樣本,每個需取(1200/40)uL。
1.5 混合樣品DNA分子的隨機斷裂
第一次使用7cycles (15on/90off,為bioruptor的400bp建議設定)。但結果發現,大部分片段散開,片段大小不集中。皆不若上次。
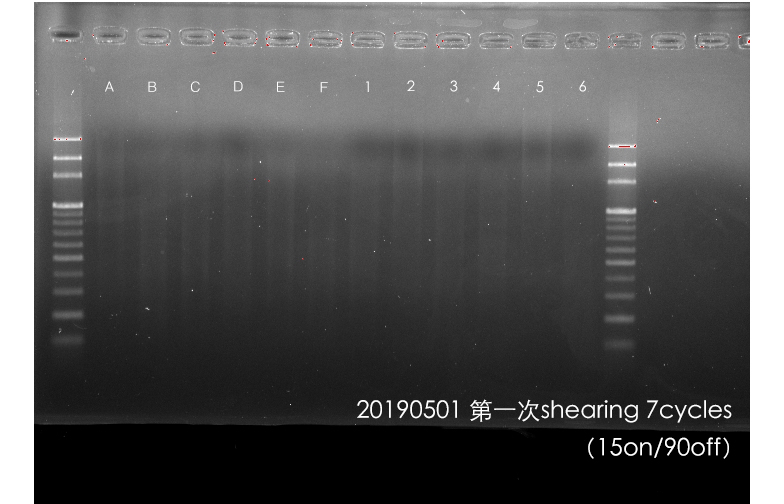
接續為4cycles ,接續上補,並確認膠圖,直至片段多集中於400~500bp為主。
最多震碎cycles為7+4+4+4+4,最少為7+4+4。

12管,由我和瓊之一人六管(為原來protocoal的總量),兩人分別繼續後續步驟。
1.6 DNA的濃縮與片段篩選
此步驟的DNA濃縮,使用Monarch® PCR & DNA Cleanup Kit (5 μg) 兩管回收,Wash buffer步驟僅使用兩次,使用水回溶,最後合併約70ul的溶液。
片段篩選後,最後回溶25uL左右,測濃度和跑膠各使用1uL,故剩餘23uL。

此步發現,>500bp的DNA片段過多(濃度大),故又另將>500bp的DNA片段震碎。並以標記「•」和原200~500bp在分管進行。

此步之後,我和瓊之各有兩管:一為原200~500bp(我:35ug/uL,瓊之:24.2ug/uL),另一為 • 200~500bp (我:18.1ug/uL,瓊之:12.2ug/uL)分管進行下面步驟。
1.7 DNA破裂端修補反應
由於總兩管總量(200~500bp、 • 200~500bp,體積為23uL)皆不足所需的1ug。故將其真空濃縮至19uL,以符合此步驟體積。
1.8 P2 adapter連結反應
此步驟共有5步:1.8 1、1.8 2、1.8 3、1.8 4、1.8 5,最後回溶22uL,測濃度和跑膠各使用1uL。(1.8 1、1.8 3、1.8 5,有留約1ul後續一起跑膠。)
片段篩選後的RAD library template濃度為:我:1.8 5:7.5ug/uL、 • 1.8 5:3.82ug/uL,瓊之:1.8 5:6.52ug/uL、 • 1.8 5:4.48ug/uL(protocol階段目標至少為:3ng/ul,最後體積為20ul,DNA量才能達50ng。)
1.9 RAD tag 擴增
1.9 1:RAD library template擴增。
我:1.8 5:7.5ug/uL,取7uL、 • 1.8 5:3.82ug/uL,取14uL,達50ng,開始擴增。
亦測試:我:1.9 1:0.436 ug/uL,瓊之:1.9 1:0.578 ug/uL。(此步體積為100ul,DNA總量為50ng。和當初放入的DNA總量一樣,並未擴增。)
1.9 2:RAD library template擴增+純化。

1.9 1 經0.7倍磁珠純化後的 1.9 2,幾乎已無DNA。